Science:利用开源计算工具预测由蛋白无序区域驱动的分子间相互作用
来源:生物谷原创 2025-09-29 13:39
该工具分析了蛋白质构建单元(称为氨基酸)的化学相互作用,并预测无序蛋白质的哪些部分会对体内的其他分子产生吸引力或排斥力。
大多数被深入研究的蛋白质都是折叠的,这意味着它们具有明确的三维形状,这有助于决定每种蛋白质的特定功能。但随着科学工具的进步,人们也认识到,许多重要的蛋白质——或蛋白质的部分——在执行关键的细胞过程时并不保持固定的形状。
这些被称为"固有无序蛋白质"的灵活结构处于持续运动状态,因此难以研究。虽然诸如获得2024年诺贝尔化学奖的AlphaFold等计算工具对于理解折叠蛋白质的工作原理具有变革性意义,但AlphaFold在研究这些柔性蛋白质方面效果不佳。更好地理解这些变形蛋白质可以识别人类疾病先前未知的原因,并为新疗法提供新思路。

如今,由华盛顿大学医学院生物化学与分子生物物理学助理教授Alex Holehouse领导并发表在《科学》杂志上的一项研究,描述了他的团队开发的一种计算方法,旨在帮助预测固有无序蛋白质的行为。
该工具分析了蛋白质构建单元(称为氨基酸)的化学相互作用,并预测无序蛋白质的哪些部分会对体内的其他分子产生吸引力或排斥力。通过仅基于化学原理进行预测,这种方法使科学家即使在缺乏明确三维结构的情况下也能模拟相互作用。
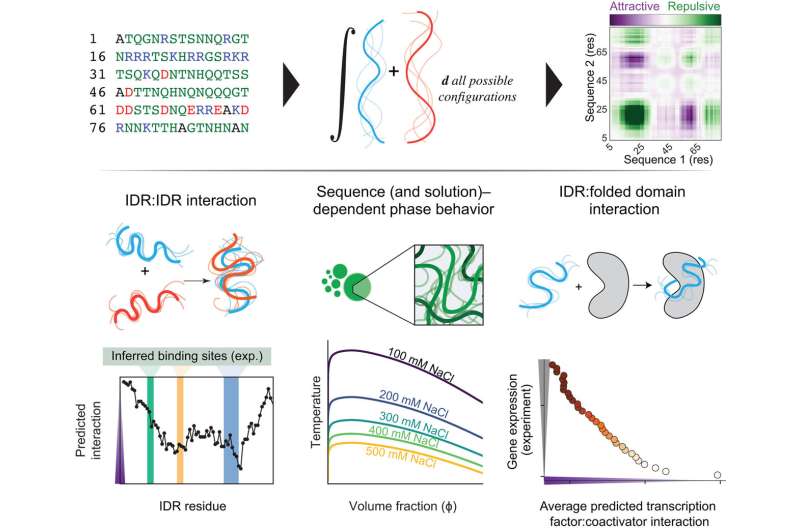
这种方法名为FINCHES,是"通过化学特异性进行第一性原理相互作用(First-principle INteractions via CHEmical Specificity)"的英文首字母缩写。它可以帮助建立关于蛋白质活性和相互作用的分子假说,这些假说随后可以在实验室中进行验证。这意味着研究人员可以开始快速探索无序蛋白质在许多关键生物学背景中可能如何工作,更重要的是,可以探索突变如何在人类疾病(尤其是癌症和神经退行性疾病)的背景下破坏蛋白质功能。
FINCHES是开源工具,研究人员可通过GitHub上的Python软件包或通过网页服务器获取。(生物谷Bioon.com)
参考文献:
Garrett M. Ginell et al, Sequence-based prediction of intermolecular interactions driven by disordered regions, Science (2025). DOI: 10.1126/science.adq8381.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


